La basura genómica ya es medicina
Casi medio centenar de científicos demuestran que los cambios genéticos asociados a 400 enfermedades no son caprichos del ADN, sino sus circuitos de regulación


Entre los muchos tesoros que guarda la 'materia oscura' del genoma, el tracional y erróneamente llamado 'ADN basura', los que tienen una utilidad más inmediata son los implicados en la enfermedad humana. La mayor parte de las variaciones hereditarias que se han asociado a enfermedades, o al riesgo de contraerlas, se hallan camufladas en esos desiertos genómicos y hasta ahora nadie sabía cómo interpretarlas. Las últimas investigaciones les han ofrecido un trabajo bien digno: regular a distancia a los genes en los tiempos y lugares adecuados. La basura genómica ha entrado por la puerta grande en la medicina.
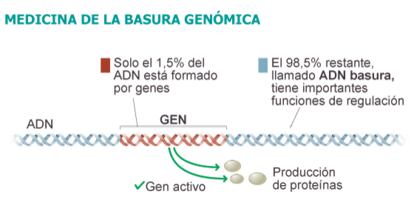
Como parte del programa Encode (acrónimo inglés de Enciclopedia de elementos de ADN), un macroproyecto de 400 científicos para descubrir y censar todos los segmentos funcionales ocultos en la 'materia oscura' del genoma --que supone el 98,5% de todo el ADN humano--, un equipo coordinado por John Stamatoyannopoulos, profesor de genómica y medicina de la Universidad de Washington, ha demostrado que la mayoría de los cambios genéticos asociados a más de 400 enfermedades no son caprichos de la basura del ADN, sino que afectan de lleno a los circuitos de regulación de los genes propiamente dichos. Presentan sus resultados en la revista 'Science'.
"Los genes solo ocupan una minúscula fracción del genoma", explica Stamatoyannopoulos, "y la mayor parte de los esfuerzos para mapear (localizar en el ADN) las causas genéticas de las enfermedades humanas se han visto frustrados por resultados que apuntaban fuera de los genes".
Los experimentos a los que se refiere Stamatoyannopoulos se llaman "estudios de asociación de amplitud genómica" ('genome-wide association studies', o GWAS en la jerga). Se han hecho varios cientos en la última década, y consisten en buscar correlaciones estadísticas entre una enfermedad, o la propensión a contraerla, y una variación en el ADN. Los estudios se hacen en poblaciones, a ser posible muy homogéneas como los mormones o los islandeses. Si las personas que sufren la enfermedad suelen llevar cierto cambio en el ADN, y las personas sanas no lo llevan, ese cambio se convierte en un sospechoso como factor causal.
La "materia oscura" del genoma supone el 98,5% de todo el ADN humano
Esta estrategia ha servido para identificar algunos genes como responsables principales de una enfermedad u otra. Si la zona del genoma que varía en la población coincide con un gen, y si ese gen interviene en procesos que están obviamente relacionados con la enfermedad en cuestión, los investigadores consiguen un argumento muy elocuente para la implicación crucial de ese gen en esa patología.
El problema es que, en la mayor parte de esos estudios de asociación, las variantes genéticas que aparecen relacionadas con la enfermedad no están en ningún gen, ni si quiera cerca de ningún gen. El grueso de toda esa variación identificada en los GWAS, en los estudios de poblaciones humanas, resulta estar 'in the middle of nowhere', en mitad de ninguna parte, como dicen los ingleses: en pleno desierto de genes, en plena 'materia oscura' del ADN, salpicada sin ton ni son por los vertederos de basura genómica. O lo que hasta ahora se consideraba basura genómica. Esto parecía dar al traste con la esperanza de encontrar los genes implicados en las principales enfermedades humanas.
Pero no es así. "Ahora", prosigue Stamatoyannopoulos, "sabemos que todos esos esfuerzos no fueron en vano, y que sus resultados estaban realmente apuntando a los 'sistemas operativos' del genoma, cuyas instrucciones están camufladas en millones de posiciones a lo largo del genoma". El científico de Washington ve los resultados de su equipo como una "nueva lente" para identificar cuál es la responsabilidad de la genética en la enfermedad.
Los estudios se hacen en poblaciones homogéneas, como los islandeses o los mormones
Las metáforas sobre las "instrucciones camufladas" y los "sistemas operativos del genoma" pueden resultar algo vaporosas, pero lo cierto es que tienen un significado muy preciso en la realidad de la genética. Los genes son 'textos', o secuencias de ADN (ttagatcgccta...) que 'significan', o determinan, otro tipo de 'texto', las secuencias de aminoácidos (LDRL...) que constituyen las proteínas, las 'nanomáquinas' en las que se basa la vida, las que permiten los flujos iónicos que transmiten la información por nuestras neuronas; las que catalizan todas las reacciones químicas que permiten a nuestras células alimentarse de energía y usarla para reconstruir sus propios componentes; las que se pegan a ciertos sitios en el ADN y modifican crucialmente sus pautas de activación.
El primer gran éxito de la biología molecular, en los mismos años sesenta en que los Beatles barrían en el show de Ed Sullivan, fue descifrar las claves de esa 'traducción' de un texto (ttagatcgccta...) en otro (LDRL...) . Cada tres letras del ADN (tta) significan un aminoácido en la proteína (L). El significado original del término 'código genético' --ya casi un arcaísmo al poco de cumplir sus 50 años-- era precisamente ese 'diccionario' para traducir el lenguaje del ADN al lenguaje de las proteínas.
Esta pieza fundamental de conocimiento, que se derivaba directamente del descubrimiento de la doble hélice del ADN en 1953 por Watson y Crick, condujo a la percepción generalizada --entre la comunidad científica-- de que los genes no eran más que esas secuencias de letras de ADN (ttagatcgccta...) que significaban secuencias de aminoácidos en las proteínas (LDRL...).
El problema de fondo es que la definición de 'gen' que hemos estado utilizando hasta ahora es demasiado simple, como ha explicado uno de los líderes del proyecto Encode, el jefe de genómica del laboratorio Cold Spring Harbor de Nueva York, un mito de la biología molecular y predio durante medio siglo de uno de los dos descubridores de la doble hélice del ADN en 1953, James Watson.
"El problema", explicó Gingeras en 2009 a este diario, "viene de muy atrás, de cuando la transmisión de la información hereditaria se veía como un problema más filosófico que bioquímico, y de los primeros trabajos sobre el ADN y sus mecanismos.; todo aquello se hizo en bacterias, que tienen unos genomas muy simples, y son los modelos con los que hemos intentado entender los genomas complejos". Sin éxito, en gran medida.
Stamatoyannopoulos y sus colegas de Washington no se han limitado a localizar ('mapear') las variaciones asociadas a enfermedades que se escondían en la basura genómica y por tanto no podían haber sido interpretadas hasta ahora. También han demostrado que, pese a estar fuera de los genes, y por lo general a enormes distancias de ellos --que pese a estar 'in the middle of nowhere', o en mitad del desierto genómico-- son los verdaderos responsables de las enfermedades de sus potadores, o de la propensión a contraerlas.
Son, después de todo, los resultados legítimos que los científicos estaban buscando. Aunque estén fuera de los genes, aunque sean 'basura', son los verdaderos culpables de buena parte de la aflicción humana. Las variaciones hereditarias camufladas en la materia oscura del genoma son las cartas marcadas, o los dados cargados, con los que nace cada individuo en la lotería genética de la que todos somos producto. Una lotería cuyo bombo, realmente, ya estaba girando en las gónadas de nuestros padres mucho antes de que nosotros fuéramos ni siquiera un proyecto.
La vanguardia de la biología cada vez tiene más de matemáticas --probablemente el destino de cualquier ciencia seria--, y los científicos de Washington han producido una notable cantidad de 'datos duros' sobre la lógica profunda de las principales enfermedades humanas. Todos ellos son sorprendentes e iluminadores. Han calculado, por ejemplo, que el 76% de las variantes genéticas 'huérfanas' --las asociadas a enfermedades en los estudios (GWAS) de la última década pero que no estaban en ningún gen-- han dado en el clavo realmente: están dentro, o al menos muy cerca, de los tramos de ADN 'basura' que no es tal basura: los que, según ha demostrado el nuevo estudio, están implicados en la regulación de los genes propiamente dichos. Es ADN regulador camuflado entre la materia oscura, una joya en la basura.
La mayoría (el 88%) de esas zonas de ADN regulador que coinciden con las variantes que habían sido asociadas a alguna enfermedad en los estudios de población están activas durante el desarrollo fetal. Casi todas estas enfermedades se revelan solo en el adulto, pero parecen tener su origen en alteraciones del desarrollo fetal.
Pero el mayor éxito del trabajo de Stamatoyannopoulos y sus colegas de Washington es que sus resultados tienen sentido biológico: la diabetes, las enfermedades metabólicas y la resistencia a la insulina, por ejemplo, convergen en los circuitos genéticos centrales para la regulación del metabolismo de la glucosa. Ha emergido del caos un concepto.
Cáncer en la avanzadilla
El descubrimiento de la utilidad de las zonas basura del genoma complica de manera extraordinaria la búsqueda de la causa genética de las enfermedades. Como ha publicado Science proyectos como el Encode (acrónimo de Enciclopedia de Elementos de ADN en inglés) van en la misma dirección. Pero, quizá, la avanzadilla esté en el proyecto ICGC (siglas en inglés de Consorcio Internacional para el Genoma del Cáncer),
en el que participa un grupo de la Universidad de Oviedo dirigido por Carlos López-Otín. Este estudia la leucemia linfocitaria crónica, unos de los ocho tumores que están en la primera fase de un trabajo que quiere llegar a establecer los mapas genómicos de unos 50 tumores.
El equipo de López Otín ya ha identificado más de 60 genes relacionados con la leucemia. Y, lo que es más preocupante (aunque interesante a la vez) es que solo uno, el Notch1, coincide en más del 15% de los pacientes. "El mapa se va complicando", ha dicho el investigador, que ha presentado este trabajo en el congreso de la Sociedad española de Bioquímica y Biología Molecular de Sevilla.
Pero nada cae en saco roto. Si cada vez está más claro que las enfermedades monogenéticas son una minoría —si no, sería demasiado fácil— en este caso por lo menos se sabe que las personas con mutaciones en ese gen van a tener un pronóstico complicado, lo que ya es una pista a la hora de establecer terapias.
En cualquier caso, sea dentro de genes tal y como los conocemos actualmente (secuencias de ADN que tienen una traducción biológica en una proteína) o en las zonas que sirven para controlarlos, todo este estudio a gran escala tiene un objetivo: encontrar las mutaciones que determinan cada enfermedad y su pronóstico. Es lo que en oncología ya se llama medicina personalizada. Se trata de una revolución que empezó caso a caso (el famosos hallazgo de los genes Brca1 o Her1 en el cáncer de mama) que ha llevado a tratamientos específicos.
Ellos fueron los primeros de una tendencia que va en aumento. Como dice un editorial de la revista Science, todo este trabajo tiene un objetivo: encontrar biomarcadores, secuencias de ADN o proteínas relacionadas que permitan detectar rápidamente que enfermedad afecta a cada uno, como la PSA del cáncer de próstata. Y ese será el primer paso para el diagnóstico y para el tratamiento.
Tu suscripción se está usando en otro dispositivo
¿Quieres añadir otro usuario a tu suscripción?
Si continúas leyendo en este dispositivo, no se podrá leer en el otro.
FlechaTu suscripción se está usando en otro dispositivo y solo puedes acceder a EL PAÍS desde un dispositivo a la vez.
Si quieres compartir tu cuenta, cambia tu suscripción a la modalidad Premium, así podrás añadir otro usuario. Cada uno accederá con su propia cuenta de email, lo que os permitirá personalizar vuestra experiencia en EL PAÍS.
¿Tienes una suscripción de empresa? Accede aquí para contratar más cuentas.
En el caso de no saber quién está usando tu cuenta, te recomendamos cambiar tu contraseña aquí.
Si decides continuar compartiendo tu cuenta, este mensaje se mostrará en tu dispositivo y en el de la otra persona que está usando tu cuenta de forma indefinida, afectando a tu experiencia de lectura. Puedes consultar aquí los términos y condiciones de la suscripción digital.




























































