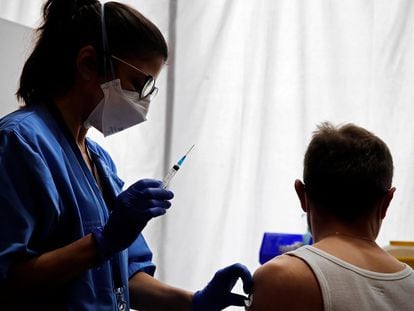
La ciencia busca sistemas de detección rápida de las nuevas variantes del coronavirus para evitar la cuarta ola
Las nuevas tecnologías permiten un seguimiento más completo de las versiones más peligrosas del coronavirus

En lo peor de la tercera ola, España solo secuenciaba el 0,078% de las muestras de pacientes infectados por coronavirus: era como intentar conocer las dimensiones de un enorme iceberg usando solo la palma de una mano. El país está poniendo ahora en marcha un sistema de vigilancia epidemiológica para analizar a sectores aleatorios de la población y conocer la prevalencia real de la variante británica y de otras también preocupantes como la brasileña o la sudafricana. Varios sistemas, algunos ya comerciales, otros en desarrollo, podrían agilizar esta tarea ante la posibilidad de una cuarta ola.
“La vigilancia epidemiológica de las nuevas variantes es más importante que nunca”Iñaki Comas, genetista
Un estudio recién publicado describe un nuevo sistema de diagnóstico rápido que podría ser parte de la solución en la lucha contra las nuevas variantes. Lo ha diseñado el equipo del investigador Juan Carlos Izpisua en el Instituto Salk de California con la idea de conseguir una forma rápida y económica de saber si alguien está infectado y con qué variante.
“Este método es barato y práctico”, explica Izpisua. “El operador no tiene que tener ninguna experiencia previa, solo meter la muestra biológica en un tubo. Podría estar en cualquier servicio de urgencias de un hospital o llevarlo a cualquier sitio, residencias, escuelas, y tener una valoración rápida y exacta de los patógenos presentes en una comunidad determinada sin ninguna infraestructura especial”, añade.
En la actualidad, los diagnósticos se hacen con una máquina PCR que tarda algo más de dos horas. Después hay que secuenciar el genoma completo del virus hallado en cada paciente. Ambos sistemas son muy fiables y aportan una gran cantidad de información.
El prototipo diseñado por el equipo de Izpisua hace un trabajo menos meticuloso, pero a cambio es muy rápido. Cabe en un maletín y consiste en un test rápido que puede analizar las muestras de 96 pacientes a la vez y decir si están contagiados en 15 minutos. El sistema lleva incorporado un secuenciador genético de bolsillo. Este aparato no lee el genoma completo del virus, que tiene 30.000 letras, sino solo partes específicas donde se encuentran las mutaciones que caracterizan a cada variante, en este caso la británica. La secuenciación lleva algo más de tres horas, con lo que el tiempo completo de análisis es de unas tres horas y media, mucho menos de lo que se tarda habitualmente usando PCR y secuenciadoras convencionales. Los detalles del sistema se han publicado en la revista Med.
Este dispositivo se sumaría a otros que ya existen en el mercado y que han sido claves en el seguimiento de nuevas variantes. Un método desarrollado por la Universidad de Oxford se ha empleado en laboratorios de móviles capaces de hacer tests rápidos en lugares remotos e incluso identificar las nuevas variantes.
La revolucionaria técnica de edición genética CRISPR también permite diagnosticar un contagio en 40 minutos. Hace apenas unos días, científicos del centro estatal de análisis genómico de Nueva Delhi, en India, anunciaron en The Lancet un nuevo método de diagnóstico que usa CRISPR y que permite detectar un contagio y la variante de interés en una hora y media y por un precio de unos 10 euros.
Un sistema desarrollado por el equipo de Juan Carlos Izpisua permite diagnosticar un contagio e identificar la variante en menos de cuatro horas
La ventaja del prototipo de Izpisua es que sería capaz de detectar también otros virus cuyos síntomas podrían confundirse con la covid, como la gripe y otros coronavirus distintos al SARS-CoV-2. “La sensibilidad es comparable a los ensayos de PCR actuales”, explica Izpisua. “La tasa estimada de falsos negativos es del 3,8% y la precisión estimada para la detección del SARS-CoV-2 es del 93%”, añade. En esta investigación también ha participado la Universidad Católica San Antonio de Murcia.
“En los países desarrollados como España la PCR no es un problema, no habría necesidad de sustituirla por sistemas como este”, explica Carmen Cámara, portavoz de la Sociedad Española de Inmunología. “Pero este tipo de instrumentos permitirían hacer un seguimiento de las variantes mucho más completo que el actual y de forma mucho más rápida, lo que es muy importante. Aquí en España tenemos que hacer un seguimiento mucho mejor que el que hacemos”, destaca. “Además, este tipo de dispositivos pueden ser fundamentales en el tercer mundo, donde no hay abundancia de PCR”, añade.
La Unión Europea recomienda que los países secuencien el genoma de al menos el 10% de todos los casos detectados por PCR, un objetivo del que España aún está lejos y que solo superan países como el Reino Unido o Dinamarca, que están haciendo un especial esfuerzo de secuenciación.
“Debido a la bajada de casos a nivel nacional ahora debemos estar ya secuenciando cerca del 5% de todos los casos positivos”, explica Iñaki Comas, codirector del consorcio español que vigila los genomas del coronavirus. Comas destaca que a pesar de la bajada de infecciones España está “en un momento muy peligroso”. “La vigilancia epidemiológica de las nuevas variantes es más importante que nunca. Aún tenemos una gran parte de la población que no está vacunada y eso reduce la presión selectiva sobre el virus y le puede dar margen para encontrar soluciones nuevas, por ejemplo mutaciones que le permitan escapar a las vacunas”, resalta.
“Este tipo de dispositivos supone un paso adelante en la aplicación de las técnicas de secuenciación masiva en el diagnóstico y caracterización de patógenos”, opina Fernando González Candelas, codirector del consorcio de secuenciación español y genetista de la Universidad de Valencia. “Funcionará mejor con virus, pero podría extenderse también a bacterias, con algunas modificaciones”, resalta.
Puedes seguir a MATERIA en Facebook, Twitter e Instagram, o apuntarte aquí para recibir nuestra newsletter semanal.
Sobre la firma

/cloudfront-eu-central-1.images.arcpublishing.com/prisa/HH6ZETAV3FD4ZHIFC6UAQYOMEQ.jpg)